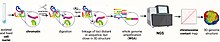
Método de captura de la conformación de los cromosomas
[2] Las frecuencias de interacción pueden ser analizadas directamente, o pueden ser convertidas a distancias y utilizadas para reconstruir estructuras 3D .
En contraste, Hi-C cuantifica interacciones entre todos los pares posibles de fragmentos simultáneamente.
Este ensayo se usaba para mostrar que el estrógeno inducía la interacción entre el promotor del gen de prolactina y un activador próximo.
ej., Giorgetti, L., Okamoto, I., Criado, N., Piolot, T., van Berkum, N.L., Meisig, J., Sedat, J., Gribnau, J., Barillot, E., Blüthgen, N., Dekker, J., Oído, E. Espacial partitioning of the regulatory landscape of the X-inactivation centre.
485(7398):381-385. https://doi.org/10.1038/nature11049 En 2018, Longzhi Tan y colaboradores investaron la técnica Dip-C.[24] Todos los métodos 3#C comienzan con unos pasos similares en una muestra de células.
[25] El tratamiento más común es: 1-3% de formaldehído durante 10-30min a temperatura ambiente, aun así, es necesario una estandarización para impedir el entrecruzamiento proteína-ADN, ya que esto puede afectar negativamente a la eficacia de digestión en el paso siguiente.
[28] Posteriormente,los loci que interaccionan se cuantifican por métodos de PCR .
Por ejemplo, 3C puede ser usado para comprobar un loci candidato de ser activador del promotor.
Los resultados obtenidos utilizando 4C son altamente reproducibles con la mayoría de las interacciones próximas que se detectan.
En concreto, se modifica el paso de ligación con biotina del método Hi-C.
Sin embargo, para resolver los haplotipos dudosos se establece un algoritmo en base a la hipótesis del vecindario.
Ésta postula que dos haplotipos homólogos deben tener diferentes patrones de contacto y que, por tanto, los haplotipos desconocidos de un cromosoma deben contactar en una región cromosómica cercana o vecina.
De este modo, se consigue establecer qué haplotipo celular está implicado en cada contacto cromosómico.
El estudio llevado con este método confirma que detecta más contactos entre los cromosomas y un menor número de falsos positivos.
Estos métodos son capaces de producir mayor sensibilidad y resolución que las basadas en 4C.
[cita requerida] ChIA-PET combina Hi-C con ChIP-seq para detectar todas las interacciones mediante una proteína de interés.
Estos métodos han revelado una organización a gran escala del genoma en dominios asociados topologicamente (TADs), los cuales se correlacionan con marcadores epigenéticos.
Estos mapas han permitido asignar genes diana para los elementos reguladores en cis (tanto potenciadores como promotores).
Además, muchos de los SNPs asociados con enfermedades y rasgos fenotípicos se localizan en estas regiones, por lo que estos mapas han permitido caracterizar genes diana para estas variantes génicas.
Esto permite entender la base molecular de enfermedades genéticas debidas a polimorfismos en zonas del ADN no codificantes.